黄学辉/戴绍军研究组合作绘制耐盐碱牧草小花碱茅高质量基因组图谱
土壤盐碱化严重影响植物生长发育,导致作物产量降低。盐碱土中的碱性盐成分(如Na2CO3和NaHCO3)不仅对植物造成离子胁迫,导致植物渗透失衡,而且可以造成pH胁迫,比中性盐(NaCl)对植物的伤害更大【1】。禾本科盐生牧草小花碱茅(Puccinellia tenuiflora),也称星星草,具有很强的耐盐碱能力,在我国东北和西北地区的盐碱地广泛分布,是苏打碱型盐碱地的先锋植物,其茎叶蛋白质含量较高,也是一种优良的牧草(图1)【2】。

图1.我国东北盐碱地生长的小花碱茅(小穗与小花)
在适应碱性盐逆境过程中,小花碱茅进化出独特的结构和代谢过程。近年来,一些小花碱茅盐碱应答基因相继被表征,如Ca2+/H+逆转运蛋白基因PutCAX1、Na+/H+逆向转运蛋白基因PutNHX、高亲和K+转运蛋白基因PutHKT2;1、K+通道蛋白基因PutAKT1、Na+/H+逆向转运蛋白基因PtNHA1、PMP3家族蛋白基因PutPMP3-1和PutPMP3-2、II型金属硫蛋白类似蛋白基因PutMT2,以及类萌发素蛋白基因PutGLP等,这些基因对于维持离子转运、离子区室化以及ROS稳态等具有重要作用【2-5】。然而,由于缺乏全基因组信息,深入的分子遗传学研究很难开展【6-7】。
近日,SCIENCE CHINA Life Sciences发表了上海师范大学黄学辉教授团队与戴绍军教授团队合作完成的题为A high-quality genome sequence of alkaligrass provides insights into halophyte stress tolerance的研究论文,报道了耐盐碱牧草小花碱茅高质量基因组序列。

黄学辉团队与戴绍军团队利用Illumina、PacBio、10X Genomics和Hi-C技术,获得了小花碱茅高质量的染色体水平的基因组序列。大约1.50 Gb的小花碱茅基因组包含38,387个蛋白质编码基因,其中54.9%为转座元件,长末端重复最为丰富。
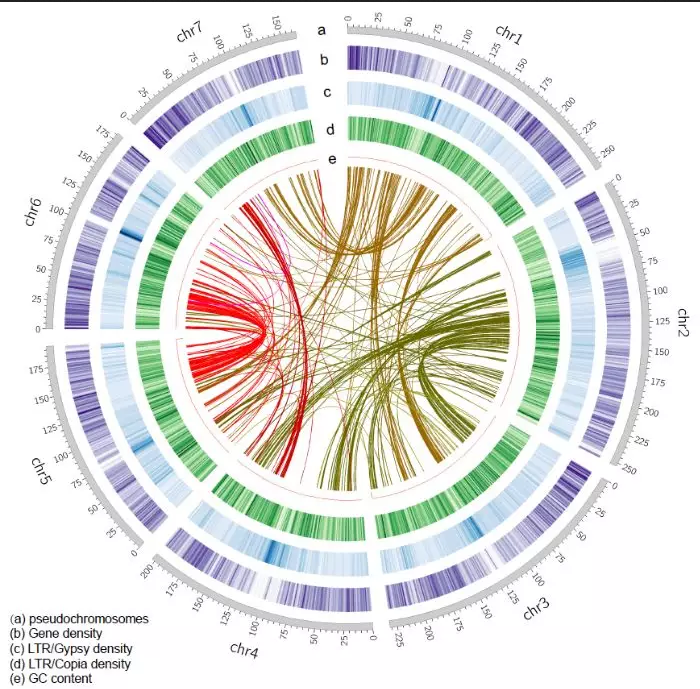
图2. 染色体级别的小花碱茅参考基因组图谱
比较基因组学与盐碱应答转录组学分析揭示了小花碱茅中大量的盐碱应答基因(图2)。其中,一系列基因的表达模式也暗示着LLG1作为分子伴侣与类受体激酶FER和FLS2等共同响应盐碱信号,调节下游的Ca2+、ABA和ROS信号通路,参与盐碱胁迫应答(图3),进一步验证这些通路在盐碱应答中的作用十分重要。
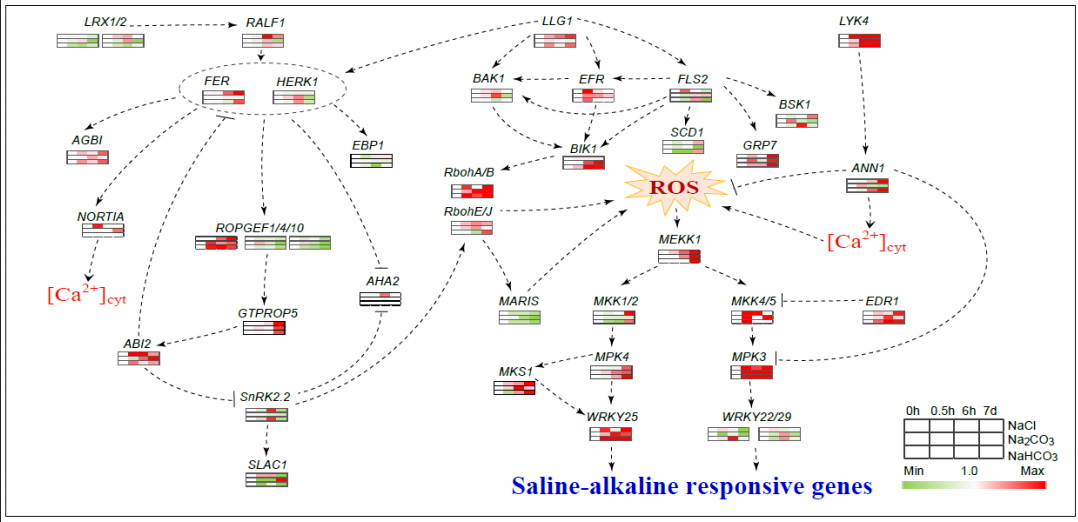
图3.小花碱茅类受体激酶FER和FLS2调控的信号通路参与盐碱应答
小花碱茅的基因组注释与盐碱应答基因的鉴定为禾本科盐生植物资源利用与进化遗传学研究提供了重要信息,也为进一步揭示单子叶盐生植物盐碱应答分子机理提供了新线索。
上海师范大学张闻婷博士和刘杰博士为共同第一作者,黄学辉教授、戴绍军教授和胡芬红博士为共同通讯作者。上海师范大学邱杰博士、东北林业大学博士生张咏雪、李莹博士和郑宝江博士参与了研究工作。
小花碱茅的基因组序列及基因注释数据下载链接:
www.xhhuanglab.cn/data/alkaligrass.html
Zhang H, Han B, Wang T, Chen S, Li H, Zhang Y, Dai S.(2012) Mechanisms of plant salt response: insights from proteomics. J Proteome Res 11(1):49-67.
Meng X, Zhao Q, Jin Y, Yu J, Yin Z, Chen S, Dai S. (2016) Chilling-responsive mechanisms in halophyte Puccinellia tenuiflora seedlings revealed from proteomics analysis. J Proteomics 2016, 143: 365-381.
Yu J, Chen S, Zhao Q, Wang T, Yang C, Diaz C, Sun G, Dai S. Physiological and proteomic analysis of salinity tolerance in Puccinellia tenuiflora. J Proteome Res 10(9):3852-70.
Yin Z., Zhang H., Zhao Q.,·Yoo M-J.,·Zhu N.,·Yu J.,·Yu J.,·Guo S.,·Miao Y.,·Chen S.,·Zhi Qin,·Dai S. Physiological and comparative proteomic analyses of saline-alkali NaHCO3-responses in leaves of halophyte Puccinellia tenuiflora. Plant and Soil, 2019, 437:137-158.
Yu J, Zhang Y, Liu J, Wang L, Liu P, Yin Z, Guo S, Ma J, Lu Z, Wang T, She Y, Miao Y, Ma L, Chen S, Li Y, Dai S. Proteomic discovery of H2O2 response in roots and functional characterization of PutGLP gene from alkaligrass. Planta 248(5):1079-1099.
Burton, J.N., Adey, A., Patwardhan, R.P., Qiu, R., Kitzman, J.O., and Shendure, J. (2013). Chromosome-scale scaffolding of de novo genome assemblies based on chromatin interactions. Nat Biotechnol 31, 1119-1125.
Chin, C.S., Peluso, P., Sedlazeck, F.J., Nattestad, M., Concepcion, G.T., Clum, A., Dunn, C., O'Malley, R., Figueroa-Balderas, R., Morales-Cruz, A., Cramer GR, Delledonne M, Luo C, Ecker JR, Cantu D, Rank DR, Schatz MC. (2016). Phased diploid genome assembly with single molecule real-time sequencing. Nat Methods 13, 1050-1054.
